kritische Zusammenfassung der Dokumentation: "Der Gen-Code" (pdf)
Roland Heynkes 25.5.2015, CC BY-SA-4.0 DE
If you cannot read German-language texts, you could try a translation into your native language with DeepL.
Navigiere mit den folgenden Links:
Auf dieser Seite fasse ich kritisch zusammen, was ich in der zweiteiligen Dokumentation: "Der Gen-Code" gesehen habe.
| Quellen |
|---|
| Der Gen-Code 1 Das Buch des Lebens |
| Der Gen-Code 2 Die Entschlüsselung des Menschen |
| animiertes Schema der DNA-Doppelhelix |
|---|

|
| Richard Wheeler, CC BY-SA 3.0 |

Unter dem Titel: "Der Gen-Code" senden seit 2014 gelegentlich die Fernsehsender GEO Television und ZDF eine verkürzte Version der BBC-Dokumentation: "The Gene Code" aus dem Jahr 2011. Darin erklärten einige Naturwissenschaftler wichtige Erkenntnisse damals moderner Genom-Forschung. Wie bei deutschen Übersetzungen angelsächsischer Wissenschaftssendungen üblich, leidet auch diese deutschsprachige Version unter den Fehlern eines Übersetzers, dessen biologische Kenntnisse nicht ausreichen. Deshalb trifft es sich gut, dass man im World Wide Web beide Folge des Zweiteilers im Original ansehen kann:

Wo auch immer auf und in unserem Planeten Biologen gründlich danach suchten - praktisch überall fanden sie Lebewesen, deren Spezies sich selbst an extremste Umweltbedingungen angepasst haben. Aber obwohl sich Archäen, Bakterien, pflanzliche, pilzliche und tierische Einzeller und Vielzeller stark von einander unterscheiden, bestehen die Baupläne in ihren Zellen alle aus DNA-Doppelsträngen. Lediglich bei einigen Viren-Spezies bestehen die Genome in der nicht aktiv lebenden Transportform aus RNA. Wirklich allen bisher daraufhin untersuchten Viren und zellulären Lebewesen gemeinsam ist aber die Sprache (die Codierung), in welcher die Baupläne geschrieben sind. Der Londoner Professor für Genetik Steve Jones schließt daraus, dass die bekannten, heute noch auf der Erde existierenden Spezies alle von nur einem gemeinsamen Urahnen abstammen ("Das Leben ist kein Wald, sondern ein Baum").
Nachdem man die Genome von Menschen und Tausenden anderen Spezies vollständig sequenziert (gelesen oder entschlüsselt) hatte, führten genaue Analysen und Vergleiche zu einer wichtigen Erkenntnis. Unsere DNA enthält nicht nur eine schockierend geringe Anzahl von Genen (nur 20-25 Tausend), sondern auch zahlreiche Spuren unserer Entwicklungsgeschichte. So zeigt beispielsweise der Vergleich mit den Genomen von Archäen und Bakterien auffällige Ähnlichkeiten zwischen DNA-Sequenzen in Archäen und unseren Zellkernen sowie zwischen heutigen Bakterien und unseren Mitochondrien. Noch heute findet man 200 Gene in Archäen und in unseren Zellkernen. Daraus schließt unter anderem der bekannte Londoner Evolutionsbiologen Dr. Nick Lane, dass wir Eukaryoten von einer Archäe abstammen, die vor 1-2 Milliarden Jahren ein Bakterium in sich aufnahm. Daraus entstand eine Endosymbiose, also eine für beide Partner vorteilhafte Partnerschaft, in der die Archäe immer größer und komplexer werden konnte, weil sie von ihren heute Mitochondrien genannten Bakterien mit chemischer Energie versorgt wurde.
Durch DNA-Sequenz-Vergleiche konnten unter anderem der in Oxford lehrende Prof. Peter Holland und die im Trinity College Dublin tätige Aoife McLysaght zeigen, dass eine Vervierfachung des Genoms eines frühen Vorfahren eines heutigen Meereswurms (des Lanzettfischchens Branchiostoma lanceolatum) vor rund einer halben Milliarde Jahren zur Entwicklung der Wirbeltiere führte.
Vergleicht man die Genome von Schlangen und Menschen, dann sieht man die gleichen Gene für die Bildung der Wirbelknochen. Statt der menschlichen 33 hat beispielsweise eine Boa Constrictor über 300 Wirbelknochen, weil bei ihr die Wirbelknochen-Gene während der Embryonalentwicklung in viel kürzeren Abständen aktiviert werden.
Der Humangenetiker Dr. Matt Hurles im Wellcome Trust Sanger Institute erklärt, dass es im menschlichen Genom nicht nur zu Austauschen einzelner Buchstaben kommt. In der menschlichen DNA-Sequenz kann man durch Vergleiche innerhalb und außerhalb unserer Spezies erkennen, dass ganze Abschnitte verloren gingen oder verdoppelt wurden. Dabei wurden Kopien teilweise weit entfernt und manchmal sogar in umgekehrter Richtung in das Genom eingefügt. Nach einer Vervielfältigung eines Gens entwickeln sich die Kopien unabhängig voneinander weiter und können unterschiedliche Funktionen annehmen.
Während der Vorherrschaft der Dinosaurier waren die Säugetiere sehr wahrscheinlich zu einer nachtaktiven oder unterirdischen Lebensweise gezwungen. Vermutlich ist das der Grund für das verglichen mit Vögeln schwach ausgeprägte Farbensehen der Säugetiere. Die meisten Säugetiere besitzen nur zwei Sorten von Farbsinneszellen (Zapfen) und sehen deshalb nur Farben, die sich aus blauem und grünem Licht mischen lassen. Vergleiche menschlicher und anderer Primaten-Genome haben gezeigt, dass es vor einigen Millionen Jahren in einem frühen Menschenaffenvorfahren auf dem X-Chromosom zu einer Verdopplung des Gens für das auf grünes Licht reagierende Lichtsinneszell-Pigment kam. Eine der beiden Kopien mutierte so, dass wir und unsere nächsten Verwandten unter den Primaten heute auch rotes Licht wahrnehmen und dadurch reife von unreifen Früchten unterscheiden können.
Die Evolution verläuft aber nicht zielgerichtet zu immer differenzierteren Sinnesleistungen. Im menschlichen Genom fand man Hunderte defekte Gene für Riechrezeptoren, die bei Mäusen noch funktionieren und diesen Tieren einen herausragend feinen Geruchssinn geben. Werden Gene nicht mehr gebraucht, dann sammeln sich in ihnen Mutationen, bis sie kein funktionsfähiges Genprodukt mehr codieren. Dann bleiben sie als sogenannte fossile Gene im Genom. Sie können aber auch durch weitere Mutationen plötzlich brauchbare neue Eiweiße oder RNAs codieren.
Durch einen Vergleich mit dem Schimpansen-Genom schaut Prof. Steve Jones in die menschliche Frühgeschichte und erkennt, dass wir Menschen aufgrund von Gen-Mutationen viele Rezeptoren unseres Geruchssinns, sehr viel Muskelkraft, unser Fell und die meisten unserer Verdauungsenzyme verloren haben. Letzteres dürfte daran liegen, dass schon der allererste Homo sapiens von seiner Mutter gekochtes und gebratenes Essen bekam. Prof. Jones meint, Menschen könnten sich im Gegensatz zu unseren Primaten-Verwandten nicht von Rohkost ernähren. Insbesondere im Hinblick auf Inuit bin ich mir da nicht so sicher. Ich bezweifle auch die Behauptung von Prof. Jones, die genetische Vielfalt sei bei Menschen viel geringer als bei Schimpansen. Unter uns Neandertaler-Homo sapiens-Mischlingen außerhalb Afrikas mag das aufgrund relativ kleiner Gruppen von Auswanderern so sein, aber in Afrika dürfte die genetische Variabilität unter den Menschen im Gegenteil größer als unter den letzten Schimpansen sein. Unter Biologen weit verbreitet und trotzdem falsch ist auch die Schlussfolgerung, die Prof. Jones aus der vermeintlich geringen genetischen Vielfalt der Spezies Mensch zieht. Das fast alle Nichtafrikaner von relativ wenigen kleinen Gruppen gemeinsamer Ahnen abzustammen scheinen, bedeutet nämlich keineswegs, dass es irgendwann vor Zigtausend Jahren außer diesen unseren Ahnen keine weiteren Menschen gegeben habe. Mit Sicherheit gab es außer unseren Ahnen noch jede Menge Afrikaner, Neanderthaler, Denisova-Menschen, Homo floresiensis und wahrscheinlich weitere, heute ausgestorbene Menschenarten oder -rassen. Und während die Einen typische Neandertaler-Allele besitzen, sind es bei Anderen Allele, die man zuerst im Denisova-Genom fand. In Afrika fand man sogar Menschen mit Allelen, die von einer bisher gänzlich unbekannten menschlichen Rasse zu stammen scheinen, von welcher anscheinend bisher noch keine fossilen Überreste gefunden wurden.
Prof. Peter Donnelly ist Mathematiker und wendete im Zentrum für Humangenetik der Universität Oxford Methoden der Statistik an, um das menschliche Genom zu untersuchen. So fand seine Arbeitsgruppe heraus, warum Menschen und Schimpansen keine fruchtbaren Nachkommen haben können. Die Ursache sind Mutationen in einem Gen (PRDM9), welches die Stellen (Recombination hotspots) festlegt, an denen während der Bildung von Geschlechtszellen das mütterliche und das väterliche Chromosom eines Chromosomenpaares Abschnitte austauschen (Crossing over), um die genetische Variabilität der Geschlechtszellen zu steigern. Stimmen diese Recombination hotspots von Mutter und Vater nicht überein, dann entstehen in deren Nachkommen bei der Bildung neuer Geschlechtszellen beim Crossing over unterschiedlich lange Chromosomen und viele Gene gehen entweder verloren oder werden verdoppelt. Deshalb könnten Nachkommen von Menschen und Schimpansen keine funktionsfähigen Geschlechtszellen bilden.
Dr. K. Thangaraj (Centre for Cellular and Molecular Biology in Hyderabad und Präsident der Indian Society of Human Genetics) erforscht Genome in verschiedenen Volksgruppen Indiens. In der DNA fand er Unterschiede, die für sehr wenig Durchmischung zwischen den indischen Kasten seit Jahrzehntausenden sprechen. So findet man (selektiv und verbreitet) bei fast allen und fast nur bei Mitgliedern der Kaste der Vaishya (Kaste der Kaufleute) eine Mutation im Gen für die Pseudocholinesterase. Die Betroffenen können die bei Operationen unter Vollnarkose eingesetzen Muskelrelaxantien (Medikamente, die Muskeln erschlaffen lassen) Mivacurium und Suxamethonium im Blutplasma nur sehr langsam abbauen. Bei ihnen führen deshalb diese Muskelrelaxantien dazu, dass nach einer Vollnarkose die Lähmung der Atemmuskulatur noch lange anhält und eine künstliche Beatmung erforderlich macht.
Man findet in Indien auch immer noch einen Volksstamm, der von einer früheren Gruppe afrikanischer Auswanderer abstammt. Nach Ansicht von Dr. Thangaraj wanderten die früheren Einwanderer in den Süden Indiens. Später verteilten sie sich auf dem gesamten indischen Subkontinent, wo man ihre Gene heute noch findet. Erst vor etwa 50.000 Jahren sollen sich genetisch davon unterscheidbare Afrikaner in Nordindien angesiedelt haben. Auch deren Nachkommen verteilten sich später in ganz Indien, aber im Gegensatz zu der ersten Gruppe zeigen sie eine größere Verwandtschaft mit den Europäern.
Natürlich ist auch in dieser Dokumentation nicht alles ganz richtig, manches sogar komplett unsinnig und irreführend.

Nur drei Monate nach der Veröffentlichung ihres berühmten Artikels: "A Structure for Deoxyribose Nucleic Acid" in "Nature 171, 737-738" wurden James D. Watson and Francis H.C. Crick im Sommer 1953 vom Physik-Professor George Gamow in einem Brief gefragt, wie der DNA-Code funktioniert. Wo andere nur eine DNA-Doppelhelix sahen, erkannte Gamow die Möglichkeit, in der Reihenfolge der Nukleobasen Erbinformationen zu speichern. Von da an wollten Naturwissenschaftler wissen, wie unsere Zellen die in der DNA gespeicherten Erbinformationen weitergeben und nutzen können. Und dazu mussten sie vor allem verstehen, wie der mit 4 Buchstaben auskommende genetische Code übersetzt (Translation) werden kann in den Aufbau von Proteinen, deren Strukturen und Funktionen von der Reihenfolge der 20 verschiedenen Aminosäuren abhängen, aus denen die Proteine bestehen. Mit einer Reihe bahnbrechender Experimente erarbeiteten Heinrich Matthaei, Marshall Nirenberg und Philip Leder 1961-1964 die Zuordnungstabelle für die Codierung einer Aminosäure-Sequenz durch eine Nukleotid-Sequenz.
|
Die Codonsonne veranschaulicht den genetischen Code. Gelesen werden die Codons von innen nach außen. Ganz außen stehen die mit 3 Buchstaben abgekürzten Namen der Aminosäuren, die durch jeweils 3 Nukleotide codiert werden. Mouagip, Public Domain |
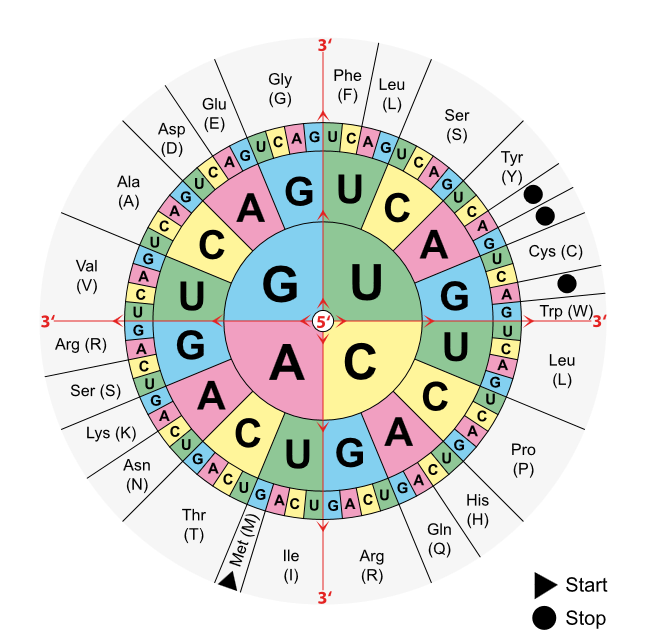 |
Die Genetik-Professorin Kay Davies gehörte zu den ersten, die nach mehr als 10 Jahren mühsamer Laborarbeit einer Erbkrankheit (Duchenne Muskeldystrophie) eine Gen-Mutation zuordnen konnten. Heute ist das in wenigen Wochen machbar. Wichtig ist das, weil die Kenntnis pathogener (krankmachender) Mutationen die Entwicklung von Gentests ermöglicht, mit denen Erbkrankheiten bei noch gesunden und sogar schon bei ungeborenen Menschen identifiziert werden können. Zunehmend werden solche Gentests während der frühen Schwangerschaft angewendet und meistens führt der Nachweis einer Erbkrankheit zu einer Abtreibung.
Heute sind Gentests für einzelne Erbkrankheiten eigentlich überflüssig. Raffinierte chemische oder physikalische Verfahren, Robotik und Computerprogramme haben ein Ausmaß von Automatisierung ermöglicht, welches die komplette Sequenzierung ganzer Genome unschlagbar günstig macht. Schon im Jahr 2011 konnte eine einzige Sequenziermaschine in einer Woche 20 menschliche Genome vollständig sequenzieren. So konnte beispielsweise Prof. Mark McCarthy in der Universität von Oxford die Genome von 3000 Diabetikern sequenzieren und fand durch den Vergleich mit gesunden Vergleichspersonen neue Mutationen in nicht codierenden Sequenzen, welche das Diabetes-Risiko erhöhen. Hat man seine eigene DNA-Sequenz auf dem eigenen Computer, dann kann man darin problemlos nach jeder heute und in naher Zukunft bekannten Erbkrankheit suchen und sogar noch unbekannte Abweichungen von gesunden Varianten unserer Gene finden. Problematisch wird es aber, wenn Andere Zugriff auf das Genom eines Menschen bekommen. Das kann heute schon zur Abtreibung eines Menschen führen. In Zukunft könnte es auch zu Problemen mit Krankenversicherungen und potentiellen Arbeitgebern führen. Sehr wahrscheinlich wird es nie eine wirklich vertrauenswürdige Institution geben, der man sein Genom zusammen mit medizinischen Informationen unbesorgt überlassen könnte. Das ist sehr schade, denn durch das Vergleichen sehr vieler menschlicher Genome ließen sich viele wertvolle Erkenntnisse für die Medizin gewinnen. Heute ist die Zahl der vergleichbaren Genome noch viel zu klein, um so gewagte Aussagen wie die angeblich besonders geringe genetische Variabilität der Spezies Mensch seriös tätigen zu können.
Das Grundprinzip der DNA-Sequenzierung nach Frederick Sanger (auch Didesoxymethode oder Kettenabbruch-Synthese genannt) ist relativ einfach. Durch kurzes Erhitzen trennt man die beiden Stränge vieler Kopien eines DNA-Doppelstranges. Dann kühlt man das Reaktionsgefäß auf eine Temperatur ab, bei der kurze Oligonukleotide (Primer) selektiv an eine komplementäre Sequenz der zu sequenzierenden DNA binden. Anschließend läßt man DNA-Polymerasen passende Nukleotide an die Starter-Oligonukleotide anhängen. In geringen Konzentrationen beigemischte fluoreszierend (früher radioaktiv) markierte Nukleotide sorgen gelegentlich für den Abbruch der Kettenverlängerung und markieren das Ende jeder neu synthetisierten Kopie der zu sequenzierenden DNA-Einzelstränge. Anschließend wird das Gemisch unterschiedlich langer, fluoreszierend markierter DNA-Fragmente gelelektrophoretisch der Größe nach getrennt. Nach einer ausreichend langen Wegstrecke werden die fluoreszierenden Nukleotide durch einen LASER-Strahl zum Leuchten angeregt und Detektoren übermitteln fortlaufend an ein aufzeichnendes Computerprogramm, mit welcher Intensität und in welchen Farben die gerade vorbeiziehenden DNA-Einzelstränge leuchten. Weil man heute die 4 Nukleotidsorten A, C, G und T mit unterschiedlich fluoreszierenden Farbstoffen markiert, lässt sich die Reihenfolge der Nukleotide direkt und sogar automatisch durch den Computer ablesen, der dann nur noch überlappende Sequenzen aus verschiedenen Sequenzierungen zu einer Gesamtsequenz zusammenfügen muss. Inzwischen wird die Sanger-Sequenzierung zunehmend durch Sequenzierungsverfahren der zweiten und dritten Generation verdrängt, die noch schneller sind und teilweise sogar die Sequenzierung der DNA und RNA einzelner Zellen ermöglichen.
| Schema der Sanger-Sequenzierung mit radioaktiver Markierung |
|---|
| Methode.svg">Dr. Norman Mauder, auf Basis einer Datei von Christoph Goemans, GNU Free Documentation License 1.2 |
| Bei radioaktiver Markierung sind vier getrennte Sequenzierungs-Reaktionen und Gelspuren erforderlich. Nur der A-Reaktion wird Didesoxyadenosintriphosphat (ddATP) für den selektiven Kettenabbruch zugegeben. In der C-Reaktion ist es Didesoxycytosintriphosphat (ddCTP) und in den G- und T-Reaktionen sind es ddGTP bzw. ddTTP. |
Nach der ersten vollständigen Sequenzierung eines menschlichen Genoms war man über die mit nur rund 20.000-25.000 verblüffend geringe Anzahl menschlicher Gene sehr überrascht. Man traute sogar seinen eigenen Daten nicht und nannte zunächst eine noch viel zu große Zahl. Zwischen den Genen fand man riesige Abschnitte (insgesamt 97-98%), die keinerlei Erbinformationen zu enthalten schienen und die deshalb sogar Junk-DNA (Schrott-DNA) genannt wurden. Diese lange an den Universitäten als vermeintliches Faktum gelehrte Meinung kann heute nur noch als Mahnung dienen, Unwissenheit nicht mit Wissen um die vermeintliche Nichtexistenz des noch Unbekannten zu verwechseln. Erkennt man in einer DNA-Sequenz keine Erbinformationen, dann ist das kein Beweis dafür, dass die DNA-Sequenz tatsächlich keine Erbinformationen enthält. Und auch wirklich Informations-freie DNA-Abschnitte könnten wichtige Funktionen haben, die man sich heute noch nicht vorstellen kann.
Heute wissen wir, dass viele Krankheiten und Eigenschaften des Menschen durch mehrere Gene, die Umwelt und das eigene Verhalten (epigenetisch) beeinflusst werden. Vor allem die letzten Jahre haben immer deutlicher gezeigt, dass wir durch ausgewogene Ernährung, ausreichend Schlaf und Erholung, ein intaktes soziales Umfeld, ein vernünftiges Maß von Ausdauersport und nicht zuletzt Muskeltrainng das Risiko von Zivilisationskrankheiten, Infektionen, Krebs und Demenz dramatisch senken können.
Vergleicht man die Ähnlichkeiten eineiiger und zweieiiger Zwillinge, dann sieht man bei vielen Eigenschaften und Krankheiten einen sehr großen Einfluss der Gene. Das gilt nicht nur für Eigenschaften wie die zu etwa 80% erblich (genetisch) bedingte Körpergröße und die Augenfarbe. Auch die Lesefähigkeit scheint zu mehr als der Hälfte erblich bedingt zu sein. Dem steht aber eine sehr viel geringere Unterschiedlichkeit der codierenden Gen-Sequenzen gegenüber. Es scheint sogar so, als würden viel mehr Eigenschaften vererbt, als wir überhaupt Gene haben. Tatsächlich beruhen die erblichen Unterschiede zwischen Menschen häufiger nicht auf unterschiedlichen Bauanleitungen, sondern auf Unterschieden hinsichtlich der Zeitpunkte und Intensitäten ihrer Benutzung. Und diese werden bestimmt durch nicht codierende Sequenzen in der vermeintichen Schrott-DNA, durch chemische Modifikationen am Gerüst der DNA (Epigenetik) und durch die räumliche Struktur der DNA. Es wird aber noch lange dauern, bis wir dieses komplexe Zusammenwirken verschiedenster Faktoren wirklich verstehen.
Natürlich ist auch in dieser Dokumentation nicht alles ganz richtig, manches sogar komplett unsinnig und irreführend.